Шкала перевода баллов на ЕГЭ 2021: минимальные баллы
Система оценивания и перевод баллов ЕГЭ вызывают много вопросов. Сколько нужно получить по каждому предмету, чтобы выдали аттестат? Что такое первичный и вторичный балл? Влияет ли оценка за экзамен на итоговую отметку в аттестате? Давайте разбираться вместе.
Минимальные баллы ЕГЭ
Выпускники, которые собираются поступать за границу, обычно выбирают для сдачи только русский язык и математику. Самым важным для них становится средний балл аттестата. Им достаточно следующих баллов:
- Русский язык — 24
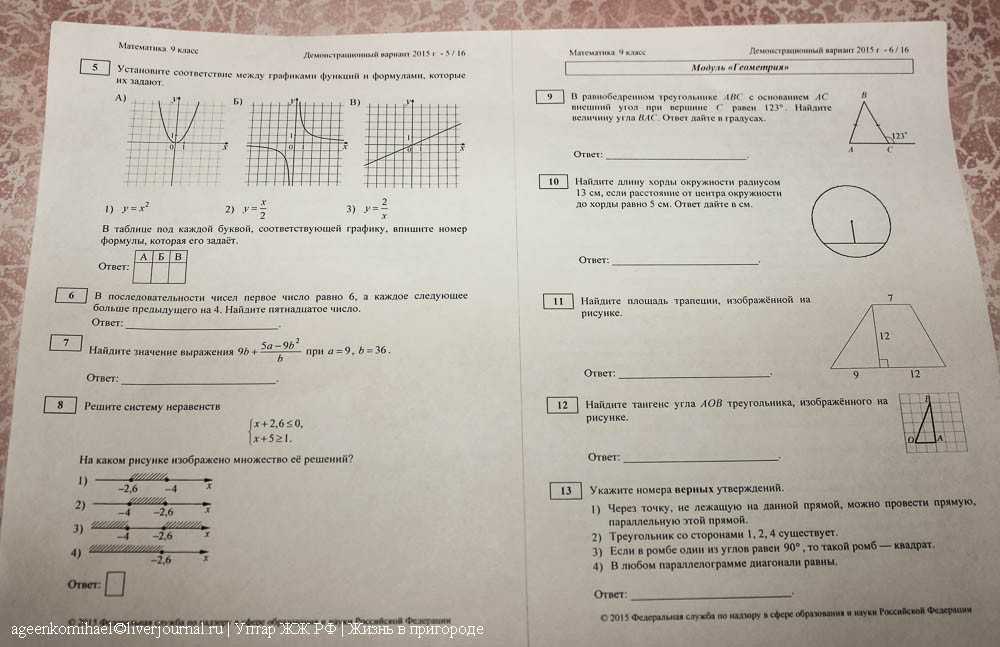
- Математика — 27
- Математика база — 3 (оценка)
А что, если не получилось уехать за границу? Можно ли поступить в российский вуз с минимальными баллами?
В 2021 году Минобрнауки установило следующий порог для поступления в вузы:
- Русский язык — 40
- Математика профильного уровня — 39
- Информатика и ИКТ — 44
- Биология — 39
- История — 35
- Химия — 39
- Иностранные языки — 30
- Физика — 39
- Обществознание — 45
- Литература — 40
- География — 40
По таким баллам в высшее учебное заведение можно поступить либо по целевому набору, либо по льготе. Балл по русскому языку должен быть выше. Кроме того, базовая математика, как результат, во вторичные баллы не переводится и при поступлении не учитывается.
Балл по русскому языку должен быть выше. Кроме того, базовая математика, как результат, во вторичные баллы не переводится и при поступлении не учитывается.
Шкала перевода баллов из первичных в стобалльные
Самое загадочное в формате ЕГЭ — перевод первичных баллов во вторичные.
Шкалирование — это процедура перевода первичных баллов в тестовые. Сперва выставляется первичный балл — это сумма баллов за все правильно выполненные задания. Первичный балл переводится во вторичный, который учитывается при поступлении в вуз.
Обычно это делается автоматически. Вместе с результатами экзамена, где указаны первичные баллы с отчётом о каждом задании (правильно/не правильно), приходят вторичные, уже переведённые в стобалльную систему. Но всегда ведь хочется знать заранее, сколько заданий нужно сделать, чтобы получить, например, 85 баллов по информатике.
Проще всего, пожалуй, с английским языком. Максимально за экзамен можно набрать 100 первичных баллов, которые автоматически превращаются во вторичные. С остальными экзаменами сложнее, потому что для каждого предмета устанавливается свое соответствие. Кроме того, в каждом экзамене есть задания, за выполнение которых первичные баллы приносят больше вторичных.
С остальными экзаменами сложнее, потому что для каждого предмета устанавливается свое соответствие. Кроме того, в каждом экзамене есть задания, за выполнение которых первичные баллы приносят больше вторичных.
Но узнать это заранее невозможно. Каждый год производится индивидуальный расчёт по каждому предмету на основе результатов работ всех выпускников, которые отсылаются в Москву и там анализируются. Тем не менее, мы составили примерную таблицу перевода первичных баллов во вторичные на 2021.
Чтобы попасть в топовые учебные заведения, такие как МГИМО или МГУ, даже ста баллов может не хватить. Поэтому будьте внимательны ко всему, что приносит дополнительные баллы — итоговому сочинению по литературе, олимпиадам, аттестату с отличием и т. д.
Влюбляем в обучение на уроках в онлайн-школе Тетрика
Оставьте заявку и получите бесплатный вводный урок
Какими будут минимальные баллы ЕГЭ в 2022
Обсуждение проекта приказа Минобрнауки будут о минимальных баллах на 2022 год.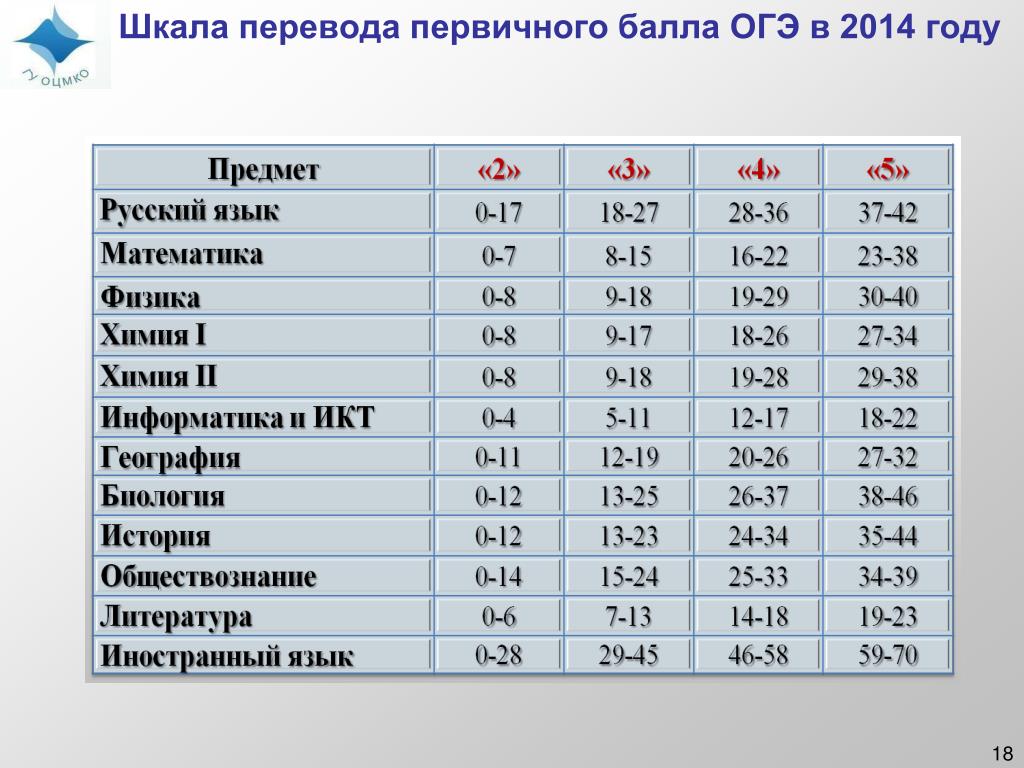
Как отмечает Евгения Матвеева – эксперт Среднерусского института управления – филиала РАНХиГС – ежегодно Министерство науки и высшего образования устанавливает минимальные баллы для поступления в вузы России. Эти баллы использует большинство учебных заведений, будут ли изменения в 2022 году?
Минобрнауки уже подготовило проект приказа, согласно которому устанавливаются минимальные баллы ЕГЭ на следующий, 2022 год. Сейчас этот документ проходит общественное обсуждение. В нём говорится: «…установить минимальное количество баллов единого государственного экзамена по общеобразовательным предметам, соответствующим специальности или направлению подготовки, по которым проводится приём на обучение в образовательных организациях, находящихся в ведении Министерства науки и высшего образования Российской Федерации, на 2022/23 учебный год согласно приложению, к настоящему приказу».
Предлагаемый на 2022 год уровень минимальных баллов Минобрнауки следующий: Русский язык – 40 баллов; Профильная математика – 39 баллов; Обществознание – 45 баллов; Физика – 39 баллов; История – 35 баллов; Биология – 39 баллов; Химия – 39 баллов; Информатика – 44 баллов; Литература – 40 баллов; География – 40 баллов; Иностранный язык – 30 баллов. На этот раз Минобрнауки оставило минимальные баллы для поступления без изменений.
На этот раз Минобрнауки оставило минимальные баллы для поступления без изменений.
В 2019 и 2018 годах порог был значительно ниже: Русский язык – 36 баллов; Профильная математика – 27 баллов; Обществознание – 42 балла; Физика – 36 баллов; История – 32 балла; Биология — 36 баллов; Химия – 36 баллов; Информатика и ИКТ – 40 баллов; Литература – 32 балла; География – 37 баллов; Иностранный язык – 22 балла.
До 2020 минимальные баллы, необходимые для поступления в вузы, подведомственные Минобрнауки, не менялись несколько лет. Минобрнауки внесло в них изменения только в 2020 году. До этого баллы, установленные Рособрнадзором, и баллы, установленные Минобрнауки, были одинаковыми. Однако затем ведомство решило самостоятельно повысить порог. Таким образом, вузы лишились возможности удерживать «минимум» на прежнем уровне. Установить планку выше они могли, но ниже – уже нет.
На основе этого можно сделать вывод, что самые распространённые минимальные баллы (баллы Минобрнауки) вполне могут закрепиться на текущем уровне до 2023 года или даже дольше. Такой порог сдачи ЕГЭ точно будет актуальным для тех, кто закончил в этом году 10 класс и собирается поступать в вуз в 2022 году, а также, возможно, для тех, кто этим летом переходит из 9 класса в старшую школу.
Такой порог сдачи ЕГЭ точно будет актуальным для тех, кто закончил в этом году 10 класс и собирается поступать в вуз в 2022 году, а также, возможно, для тех, кто этим летом переходит из 9 класса в старшую школу.
Минимальные баллы Минобрнауки действуют только для вузов, которые подчиняются этому ведомству. И таких – большинство. Однако существуют вузы, подведомственные другим министерствам. Там минимальные баллы часто бывают ниже – вплоть до уровня прошлых лет, аналогичного установленному уже Роспотребнадзором.
Назад
АДМИНИСТРАЦИЯ
КРОМСКОГО РАЙОНА
официальный сайт
2023, Все права защищены
+7 (48643) 2-29-04
п. Кромы, пл. Освобождения, д.1
Создание сайтов
Настройка порога стоимости для параллелизма Параметр конфигурации сервера — SQL Server
Твиттер LinkedIn Фейсбук Электронная почта
- Статья
- 3 минуты на чтение
Применяется к: SQL Server
В этом разделе описывается настройка порога стоимости для параллелизма 9Параметр конфигурации сервера 0020 в SQL Server с помощью SQL Server Management Studio или Transact-SQL. Параметр порога стоимости 
В этом разделе
Прежде чем начать:
Ограничения и запреты
Рекомендации
Безопасность
Чтобы настроить порог стоимости для опции параллелизма, используйте:
Студия управления SQL Server
Transact-SQL
Последующие действия: После настройки порога стоимости для параметра параллелизма
Перед началом работы
Ограничения и запреты
Стоимость относится к абстрагированной единице стоимости, а не к единице расчетного времени. Установите порог стоимости
SQL Server игнорирует порог стоимости для значения параллелизма при следующих условиях:
Ваш компьютер имеет только один логический процессор.
Для SQL Server доступен только один логический процессор из-за параметра конфигурации affinity mask .

Для параметра максимальная степень параллелизма установлено значение 1.
Логический процессор — это базовая аппаратная единица процессора, которая позволяет операционной системе диспетчеризировать задачу или выполнять контекст потока. Каждый логический процессор может одновременно выполнять только один контекст потока. Ядро процессора — это схема, обеспечивающая возможность декодирования и выполнения инструкций. Ядро процессора может содержать один или несколько логических процессоров. Следующий запрос Transact-SQL можно использовать для получения информации о ЦП для системы.
ВЫБЕРИТЕ (cpu_count / hyperthread_ratio) AS PhysicalCPUs, cpu_count КАК логические ЦП ИЗ sys.dm_os_sys_info
Рекомендации
Этот параметр является дополнительным и должен изменяться только опытным администратором базы данных или сертифицированным специалистом по SQL Server.
В некоторых случаях может быть выбран параллельный план, даже если план стоимости запроса меньше текущего порога стоимости для значения параллелизма .
 Это может произойти из-за того, что решение об использовании параллельного или последовательного плана основано на оценке затрат, предоставленной ранее в процессе оптимизации. Дополнительные сведения см. в Руководстве по архитектуре обработки запросов.
Это может произойти из-за того, что решение об использовании параллельного или последовательного плана основано на оценке затрат, предоставленной ранее в процессе оптимизации. Дополнительные сведения см. в Руководстве по архитектуре обработки запросов.Хотя значение по умолчанию 5 подходит для большинства систем, может подойти и другое значение. Выполните тестирование приложения с более высокими и более низкими значениями, если это необходимо для оптимизации производительности приложения.
Безопасность
Разрешения
Разрешения на выполнение в sp_configure без параметров или только с первым параметром предоставляются всем пользователям по умолчанию. Чтобы выполнить sp_configure с обоими параметрами для изменения параметра конфигурации или для запуска оператора RECONFIGURE, пользователю необходимо предоставить разрешение ALTER SETTINGS на уровне сервера. Разрешение ALTER SETTINGS неявно принадлежит sysadmin и serveradmin фиксированные роли сервера.
Использование SQL Server Management Studio
Настройка порога стоимости для параметра параллелизма
В обозревателе объектов щелкните сервер правой кнопкой мыши и выберите Свойства .
Щелкните узел Advanced .
В разделе Parallelism измените параметр Cost Threshold for Parallelism на нужное значение. Введите или выберите значение от 0 до 32767.
Использование Transact-SQL
Чтобы настроить порог стоимости для параметра параллелизма
Подключитесь к компоненту Database Engine.
На стандартной панели щелкните
Скопируйте и вставьте следующий пример в окно запроса и нажмите Выполнить . В этом примере показано, как использовать хранимую процедуру sp_configure для установки порогового значения стоимости
для параметра параллелизмаравным 9. 0183 10 .
0183 10 .
ИСПОЛЬЗОВАТЬ AdventureWorks2012 ; ИДТИ EXEC sp_configure 'показать дополнительные параметры', 1 ; ИДТИ РЕКОНФИГУРИРОВАТЬ ИДТИ EXEC sp_configure 'порог стоимости параллелизма', 10 ; ИДТИ РЕКОНФИГУРИРОВАТЬ ИДТИ
Дополнительные сведения см. в разделе Параметры конфигурации сервера (SQL Server).
Дальнейшие действия: После настройки порога стоимости для параметра параллелизма
Параметр вступает в силу немедленно, без перезапуска сервера.
См. также
Настройка параллельных операций с индексами
Подсказки запроса (Transact-SQL)
ALTER WORKLOAD GROUP (Transact-SQL)
affinity mask Параметр конфигурации сервера
RECONFIGURE (Transact-SQL)
Параметры конфигурации сервера (SQL Server)
sp_configure (Transact-SQL) )
Обратная связь
Просмотреть все отзывы о странице
KofamKOALA: назначение KEGG Ortholog на основе профиля HMM и порога адаптивной оценки | Биоинформатика
Журнальная статья
Такуя Арамаки,
Такуя Арамаки
Ищите другие работы этого автора на:
Оксфордский академический
пабмед
Google Scholar
Ромен Блан-Матье,
Ромен Блан-Матье
Ищите другие работы этого автора на:
Оксфордский академический
пабмед
Google Scholar
Хисаси Эндо,
Хисаси Эндо
Ищите другие работы этого автора на:
Оксфордский академический
пабмед
Google Scholar
Коити Окубо,
Коити Окубо
Ищите другие работы этого автора на:
Оксфордский академический
пабмед
Google Scholar
Минору Канехиса,
Минору Канехиса
Ищите другие работы этого автора на:
Оксфордский академический
пабмед
Google Scholar
Сусуму Гото,
Сусуму Гото
Ищите другие работы этого автора на:
Оксфордский академический
пабмед
Google Scholar
Хироюки Огата
Хироюки Огата
Ищите другие работы этого автора на:
Оксфордский академический
пабмед
Google Scholar
Биоинформатика , том 36, выпуск 7, 1 апреля 2020 г. , страницы 2251–2252, https://doi.org/10.1093/bioinformatics/btz859
, страницы 2251–2252, https://doi.org/10.1093/bioinformatics/btz859
Опубликовано:
19 ноября 2019 г.
История статьи
Получен:
09 апреля 2019 г.
Полученная ревизия:
02 октября 2019 г.
Принято:
16 ноября 2019 г.
Опубликовано:
19 ноября 2019
Фильтр поиска панели навигации БиоинформатикаЭтот выпускЖурналы по биоинформатикеБиоинформатика и вычислительная биологияКнигиЖурналыOxford Academic Мобильный телефон Введите поисковый запрос
Закрыть
Фильтр поиска панели навигации БиоинформатикаЭтот выпускЖурналы по биоинформатикеБиоинформатика и вычислительная биологияКнигиЖурналыOxford Academic Введите поисковый запрос
Advanced Search
Abstract
Summary
KofamKOALA — это веб-сервер для назначения ортологов KEGG (KO) белковым последовательностям путем гомологического поиска в базе данных скрытых марковских моделей профиля (KOfam) с предварительно вычисленными адаптивными пороговыми значениями. KofamKOALA работает быстрее, чем существующие инструменты назначения KO, а его точность сопоставима с лучшими инструментами. Аннотации функций от KofamKOALA помогают связывать гены с ресурсами KEGG, такими как карты путей KEGG, и облегчают реконструкцию молекулярной сети.
KofamKOALA работает быстрее, чем существующие инструменты назначения KO, а его точность сопоставима с лучшими инструментами. Аннотации функций от KofamKOALA помогают связывать гены с ресурсами KEGG, такими как карты путей KEGG, и облегчают реконструкцию молекулярной сети.
Доступность и внедрение
KofamKOALA, KofamScan и KOfam находятся в свободном доступе на сайте GenomeNet (https://www.genome.jp/tools/kofamkoala/).
Дополнительная информация
Дополнительные данные доступны по адресу Биоинформатика онлайн.
1 Введение
Автоматическое аннотирование функций генов является важным первым шагом в интерпретации геномных данных. Киотская энциклопедия генов и геномов (KEGG) является широко используемой справочной базой знаний, которая помогает исследовать геномные функции, связывая гены с биологическими знаниями, такими как метаболические пути и молекулярные сети (Kanehisa and Goto, 2000). В KEGG база данных KEGG Orthology (KO) — большая коллекция семейств белков (т. 0301 K идентификаторы номеров. В настоящее время KO присвоены 12 934 525 (48%) белковым последовательностям в базе данных KEGG GENES (27 173 868 белков). Три существующих инструмента, BlastKOALA, GhostKOALA (Kanehisa et al. , 2016) и KAAS (Moriya et al. , 2007), доступны для присвоения KO белковым последовательностям. Эти инструменты используют программное обеспечение для поиска гомологии, такое как BLAST (Altschul et al. , 1997) и GHOSTX (Suzuki et al. , 2014), для поиска аминокислотных последовательностей в отношении GENES. Чтобы сократить время вычислений, необходимое для множественных попарных сравнений последовательностей, эти инструменты используют выбранные репрезентативные последовательности из GENES для создания своей целевой базы данных. В этом исследовании мы предлагаем использовать скрытую марковскую модель профиля (pHMM) для сжатия базы данных и определения адаптивных порогов для оценок сходства, которые можно использовать для надежных назначений KO.
0301 K идентификаторы номеров. В настоящее время KO присвоены 12 934 525 (48%) белковым последовательностям в базе данных KEGG GENES (27 173 868 белков). Три существующих инструмента, BlastKOALA, GhostKOALA (Kanehisa et al. , 2016) и KAAS (Moriya et al. , 2007), доступны для присвоения KO белковым последовательностям. Эти инструменты используют программное обеспечение для поиска гомологии, такое как BLAST (Altschul et al. , 1997) и GHOSTX (Suzuki et al. , 2014), для поиска аминокислотных последовательностей в отношении GENES. Чтобы сократить время вычислений, необходимое для множественных попарных сравнений последовательностей, эти инструменты используют выбранные репрезентативные последовательности из GENES для создания своей целевой базы данных. В этом исследовании мы предлагаем использовать скрытую марковскую модель профиля (pHMM) для сжатия базы данных и определения адаптивных порогов для оценок сходства, которые можно использовать для надежных назначений KO.
2 Реализация
Для каждого набора белковых последовательностей в GENES, аннотированных данным KO, мы генерируем pHMM следующим образом. Во-первых, избыточность последовательностей в наборе последовательностей уменьшается с помощью CD-HIT (Fu et al. , 2012) с отсечением кластеризации 100% идентичности последовательностей. Затем для выравнивания последовательностей и создания pHMM соответственно используют MAFFT (Katoh et al. , 2005) и HMMER/hmmbuild (Eddy, 2008).
Порог адаптивной оценки для каждой семьи рассчитывается для каждой семьи с нокаутом следующим образом. Для данного семейства КО неизбыточные последовательности, принадлежащие семейству, случайным образом делятся на три группы. Одна из групп используется в качестве положительного обучающего набора данных, а последовательности в оставшихся двух группах используются для создания pHMM. Последовательности, принадлежащие остальным семействам КО, служат отрицательным обучающим набором данных для рассматриваемого КО.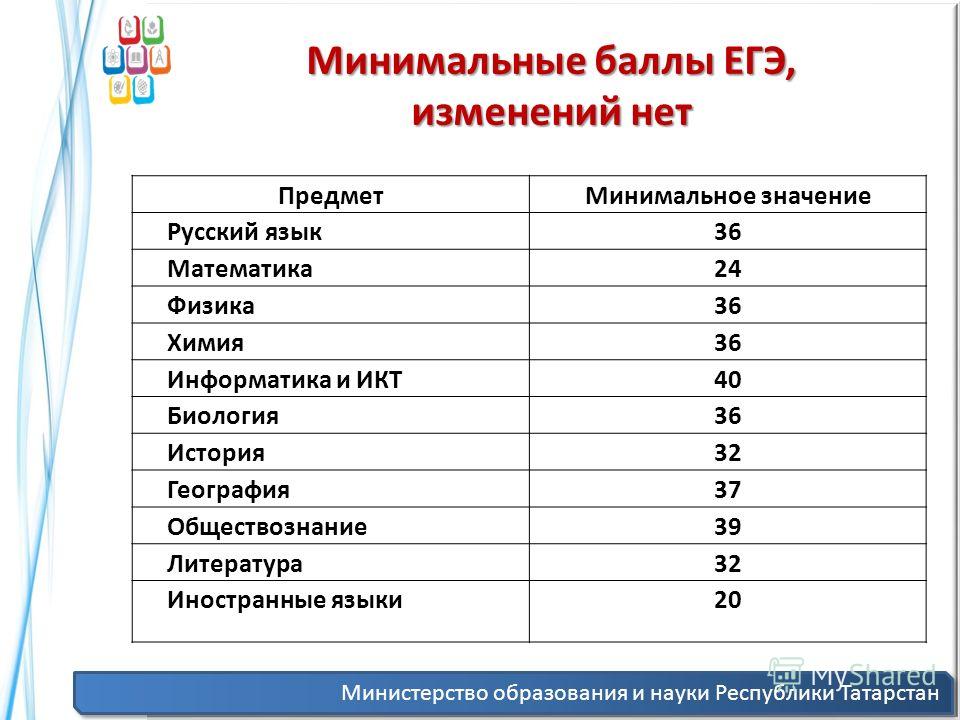 Оценки сходства последовательностей (битовые оценки) между последовательностями в положительных/отрицательных обучающих наборах данных и pHMM вычисляются с использованием HMMER/hmmsearch. Основываясь на распределениях двух наборов оценок битов для последовательностей в положительных и отрицательных наборах данных, мы определяем пороговую оценку, T , который максимизирует меру F (дополнительные данные). Эта процедура повторяется три раза путем замены положительного обучающего набора данных среди трех групп. Наконец, адаптивный порог оценки для семейства KO определяется как среднее значение T (T¯). T¯ используется в качестве критерия для присвоения семейства KO новым последовательностям.
Оценки сходства последовательностей (битовые оценки) между последовательностями в положительных/отрицательных обучающих наборах данных и pHMM вычисляются с использованием HMMER/hmmsearch. Основываясь на распределениях двух наборов оценок битов для последовательностей в положительных и отрицательных наборах данных, мы определяем пороговую оценку, T , который максимизирует меру F (дополнительные данные). Эта процедура повторяется три раза путем замены положительного обучающего набора данных среди трех групп. Наконец, адаптивный порог оценки для семейства KO определяется как среднее значение T (T¯). T¯ используется в качестве критерия для присвоения семейства KO новым последовательностям.
База данных HMM с адаптивными пороговыми значениями баллов была названа KOfam. Когда настоящее исследование проводилось с использованием KEGG версии 88.2, KOfam содержал 20 654 pHMM. Мы разработали программное обеспечение KofamScan и использовали его на веб-сервере KofamKOALA, чтобы аннотировать гены с помощью KOfam и связать их с другими ресурсами KEGG через K номера для универсального функционального исследования.
3 Оценка и обсуждение
Чтобы сравнить производительность KofamScan с BlastKOALA, GhostKOALA и KAAS, мы использовали 40 геномов (20 эукариот и 20 прокариот; дополнительная таблица S1), случайно выбранных из 6030 геномов, записанных в GENES, в качестве тестовых запросов. Этот тестовый набор содержит 383 202 последовательности (143 662 последовательности с нокаутами), что соответствует 16 166 различным нокаутам. Из GENES мы удалили все геномы, принадлежащие родам выбранных 40 геномов тестового запроса. Затем, используя оставшиеся последовательности GENES с аннотациями KO, мы создали тестовую базу данных KOfam для этой оценки. Что касается BlastKOALA, GhostKOALA и KAAS, мы использовали целевые базы данных по умолчанию, используемые на их соответствующих веб-серверах, после удаления геномов из родов, которые были представлены тестовыми запросами.
База данных KOfam, созданная для этой оценки, содержала 20 394 pHMM, из которых 9414 были представлены последовательностями прокариот. Для 40 геномов, составляющих наш тестовый набор, производительность ( F -мера) была сопоставима для KofamScan (0,866), BlastKOALA (0,889) и GhostKOALA (0,862), в то время как KAAS показал более низкую F -мера (0,810) ( Таблица 1). Чтобы выполнить еще один тест, используя только 20 прокариотических геномов в качестве тестовых запросов, мы сократили целевые базы данных, либо исключив pHMM, состоящие исключительно из эукариотических последовательностей (для KofamScan), либо используя целевую базу данных для прокариот (для BlastKOALA, GhostKOALA и KAAS). И снова производительность КофамСкан ( F = 0,875) был сравним с BlastKOALA (0,846) и GhostKOALA (0,886), в то время как KAAS показал самый низкий показатель F (0,786).
Для 40 геномов, составляющих наш тестовый набор, производительность ( F -мера) была сопоставима для KofamScan (0,866), BlastKOALA (0,889) и GhostKOALA (0,862), в то время как KAAS показал более низкую F -мера (0,810) ( Таблица 1). Чтобы выполнить еще один тест, используя только 20 прокариотических геномов в качестве тестовых запросов, мы сократили целевые базы данных, либо исключив pHMM, состоящие исключительно из эукариотических последовательностей (для KofamScan), либо используя целевую базу данных для прокариот (для BlastKOALA, GhostKOALA и KAAS). И снова производительность КофамСкан ( F = 0,875) был сравним с BlastKOALA (0,846) и GhostKOALA (0,886), в то время как KAAS показал самый низкий показатель F (0,786).
Таблица 1.
Сравнение производительности KofamScan с другими инструментами
| . | КофамСкан . | ВзрывКоала
. | ПризракКОАЛА . | КААС . |
|---|---|---|---|---|
| Entire database (40 genomes) | ||||
| Precision | 0.844 | 0.835 | 0.787 | 0.895 |
| Recall | 0.888 | 0.950 | 0.952 | 0.739 |
| F | 0,866 | 0,889 | 0,862 | 0,810 |
| База данных прокариот (20 геномов) | 9047610477 | 0.906 | 0.906 | 0.907 | 0.881 |
| Recall | 0.846 | 0.793 | 0.867 | 0.709 |
| F | 0.875 | 0.846 | 0.886 | 0.786 |
| . | КофамСкан . | ВзрывКоала
. | ПризракКОАЛА . | КААС . |
|---|---|---|---|---|
| Entire database (40 genomes) | ||||
| Precision | 0.844 | 0.835 | 0.787 | 0.895 |
| Recall | 0.888 | 0.950 | 0.952 | 0.739 |
| F | 0,866 | 0,889 | 0,862 | 0,810 |
| Prokaryote database (20 genomes) | ||||
| Precision | 0.906 | 0.906 | 0.907 | 0.881 |
| Recall | 0.846 | 0.793 | 0.867 | 0.709 |
| F | 0,875 | 0,846 | 0,886 | 0,786 |
Открыть в новой вкладке
Таблица 1.
Сравнение производительности KofamScan с другими инструментами
| . | КофамСкан . | ВзрывКоала . | ПризракКОАЛА . | КААС . |
|---|---|---|---|---|
| Полная база данных (40 геномов) | ||||
| 0480 0.895 | ||||
| Recall | 0.888 | 0.950 | 0.952 | 0.739 |
| F | 0.866 | 0.889 | 0.862 | 0.810 |
| Prokaryote database (20 genomes) | ||||
| Precision | 0.906 | 0.906 | 0.907 | 0.881 |
| Recall | 0.846 | 0.793 | 0.867 | 0.709 |
| F | 0.875 | 0.846 | 0. 886 886 | 0.786 |
| . | КофамСкан . | ВзрывКоала . | ПризракКОАЛА . | КААС . |
|---|---|---|---|---|
| Вся база данных (40 геномов) | ||||
| Precision | 0.844 | 0.835 | 0.787 | 0.895 |
| Recall | 0.888 | 0.950 | 0.952 | 0.739 |
| F | 0.866 | 0.889 | 0.862 | 0,810 |
| База данных прокариот (20 геномов) | ||||
| 07 | 0.881 | |||
| Recall | 0.846 | 0.793 | 0.867 | 0.709 |
| F | 0.875 | 0.846 | 0.886 | 0. 786 786 |
Открыть в новой вкладке
Что касается времени ЦП, KofamScan был в 69, 2,1 и 1,1 раза быстрее, чем BlastKOALA, KAAS и GhostKOALA соответственно, когда они были протестированы на аннотацию 40 геномов (дополнительные таблицы S2 и S3). Процессорное время для этого расчета составило 2 ч36 м18 с для KofamScan, в то время как для BlastKOALA оно превысило 168 ч. Для теста с 20 геномами прокариот КофамСкан составил 83, 1,9и в 1,8 раза быстрее, чем BlastKOALA, KAAS и GhostKOALA соответственно. Требуемое процессорное время для KofamScan составило 11 м59 с, а для BlastKOALA — более 16 ч. Последний результат указывает на то, что KofamScan может получить больше преимуществ от сокращения целевой базы данных по сравнению с тремя другими инструментами, хотя он входит в число инструментов, демонстрирующих самые высокие показатели F .
Мы разработали базу данных pHMM на основе баз данных KO и GENES. Пороговые значения адаптивной оценки предварительно вычисляются для отдельных семейств KO и могут использоваться для назначения KO ( K номера) в последовательности с использованием KofamScan и KofamKOALA. Совпадения последовательностей с оценкой выше порогового значения считаются более надежными, чем другие совпадения, и поэтому в результатах этих инструментов отмечаются отметками «*». Пользователи KofamScan могут настраивать KOfam, выбирая подмножество KO, чтобы они могли сосредоточиться на аннотации определенного класса белков, сокращая время вычислений. Веб-сервер KofamKOALA имеет дополнительные функции для автоматической отправки результатов поиска в KEGG Mapper для реконструкции путей (ПУТЬ), модулей путей (МОДУЛЬ) и классификации иерархических функций (BRITE).
Совпадения последовательностей с оценкой выше порогового значения считаются более надежными, чем другие совпадения, и поэтому в результатах этих инструментов отмечаются отметками «*». Пользователи KofamScan могут настраивать KOfam, выбирая подмножество KO, чтобы они могли сосредоточиться на аннотации определенного класса белков, сокращая время вычислений. Веб-сервер KofamKOALA имеет дополнительные функции для автоматической отправки результатов поиска в KEGG Mapper для реконструкции путей (ПУТЬ), модулей путей (МОДУЛЬ) и классификации иерархических функций (BRITE).
Благодарности
Время расчета предоставлено суперкомпьютерной системой Института химических исследований Киотского университета.
Финансирование
Работа выполнена при поддержке JSPS/MEXT/KAKENHI (№ 26430184, 18H02279, 16H06429, 16K21723, 16H06437) и Программы совместных исследований Института химических исследований Киотского университета (№ 2018-3 ).
Конфликт интересов : не объявлено.
Каталожные номера
Альтшул
С.Ф.
и др. (
1997
)
Gapped BLAST и PSI-BLAST: новое поколение программ поиска белков в базе данных
.
Рез. нуклеиновых кислот
.,
25
,
3389
–
3402
.
Эдди
С.Р.
(
2008
)
Вероятностная модель локального выравнивания последовательностей, упрощающая оценку статистической значимости
.
Вычисл. PLoS. Биол
. ,
,
4
,
е1000069
.
Fu
L.
и др. (
2012
)
CD-HIT: ускорено для кластеризации данных секвенирования следующего поколения
.
Биоинформатика.
Канехиса
М.
,
Перейти к
С.
(
2000
)
KEGG: Киотская энциклопедия генов и геномов
.
Рез. нуклеиновых кислот
.,
28
,
27
–
30
.
Канехиса
М.
и др. (
(
2016
)
BlastKOALA и GhostKOALA: инструменты KEGG для функциональной характеристики последовательностей генома и метагенома
.
Дж. Мол. Биол
.,
428
,
726
–
731
.
Катох
К.
и др. (
2005
)
MAFFT версии 5: повышение точности множественного выравнивания последовательностей
.
Рез. нуклеиновых кислот
.,
33
,
511
–
518
.
Мория
Ю.
и др. (
(
2007
)
KAAS: сервер автоматической аннотации генома и реконструкции путей
.
Нуклеиновые кислоты Res
.,
35
,
W182
–
W185
.
Suzuki
S.
и др. (
2014
)
GHOSTX: улучшенный алгоритм поиска гомологии последовательностей с использованием массива суффиксов запроса и массива суффиксов базы данных
.
PLoS One
,
9
,
e103833
.
© Автор(ы), 2019. Опубликовано Oxford University Press.
Это статья в открытом доступе, распространяемая в соответствии с условиями некоммерческой лицензии Creative Commons Attribution (http://creativecommons. org/licenses/by-nc/4.0/), которая разрешает некоммерческое повторное использование, распространение , а также воспроизведение на любом носителе при условии правильного цитирования оригинальной работы. По вопросам коммерческого повторного использования обращайтесь по адресу [email protected]
org/licenses/by-nc/4.0/), которая разрешает некоммерческое повторное использование, распространение , а также воспроизведение на любом носителе при условии правильного цитирования оригинальной работы. По вопросам коммерческого повторного использования обращайтесь по адресу [email protected]
Раздел выдачи:
ГЕНОМНЫЙ АНАЛИЗ
Помощник редактора: Альфонсо Валенсия
Альфонсо Валенсия
Ассоциированный редактор
Ищите другие работы этого автора на:
Оксфордский академический
пабмед
Google Scholar
Скачать все слайды
Дополнительные данные
Дополнительные данные
btz859_Supplementary_Data — zip файл
Реклама
Цитаты
Просмотры
14 793
Альтметрика- 8
Дополнительная информация о метриках
Последний
Самые читаемые
Самые цитируемые
Оповещения по электронной почте
Оповещение об активности статьи
Предварительные уведомления о статьях
Оповещение о новой проблеме
Оповещение о текущей проблеме
Получайте эксклюзивные предложения и обновления от Oxford Academic
Ссылки на статьи по телефону
Не найдено совпадений для настроенного запроса.


 Это может произойти из-за того, что решение об использовании параллельного или последовательного плана основано на оценке затрат, предоставленной ранее в процессе оптимизации. Дополнительные сведения см. в Руководстве по архитектуре обработки запросов.
Это может произойти из-за того, что решение об использовании параллельного или последовательного плана основано на оценке затрат, предоставленной ранее в процессе оптимизации. Дополнительные сведения см. в Руководстве по архитектуре обработки запросов.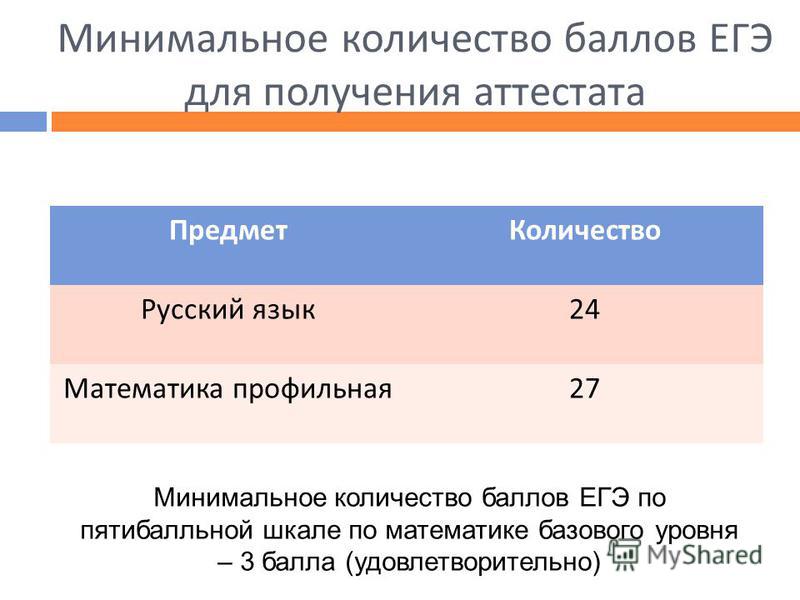 0183 10 .
0183 10 . , страницы 2251–2252, https://doi.org/10.1093/bioinformatics/btz859.
, страницы 2251–2252, https://doi.org/10.1093/bioinformatics/btz859.
Leave A Comment